2024年1月7日,南方科技大学前沿生物技术研究院朱健康院士课题组题为“An engineered Cas12i nuclease that is an efficient genome editing tool in animals and plants”的研究论文在国际著名期刊The Innovation在线发表。该研究基于AI预测Cas12i3与核酸互作位点,并通过对Cas核酸识别区域候选点突进行组合获得了高编辑活性的优化版本Cas-SF01,为疾病治疗和动植物基因编辑提供高效新工具(图1)。
近十年来,CRISPR-Cas基因编辑系统快速发展,已在基础研究、动植物育种、核酸检测和基因治疗等领域被广泛应用。Cas蛋白是基因编辑系统的核心元件,除了II型的Cas9,包括V-I 型的Cas12i家族蛋白等其他类型的Cas蛋白也相继被鉴定和解析。相较于Cas9,Cas12i蛋白尺寸相对更小,可以加工crRNA,能识别富含AT的PAM序列。自然存在的Cas蛋白普遍存在效率低,识别范围小等缺点,而通过蛋白质进化及改造可实现Cas蛋白的活性提升,提升其实际应用价值。
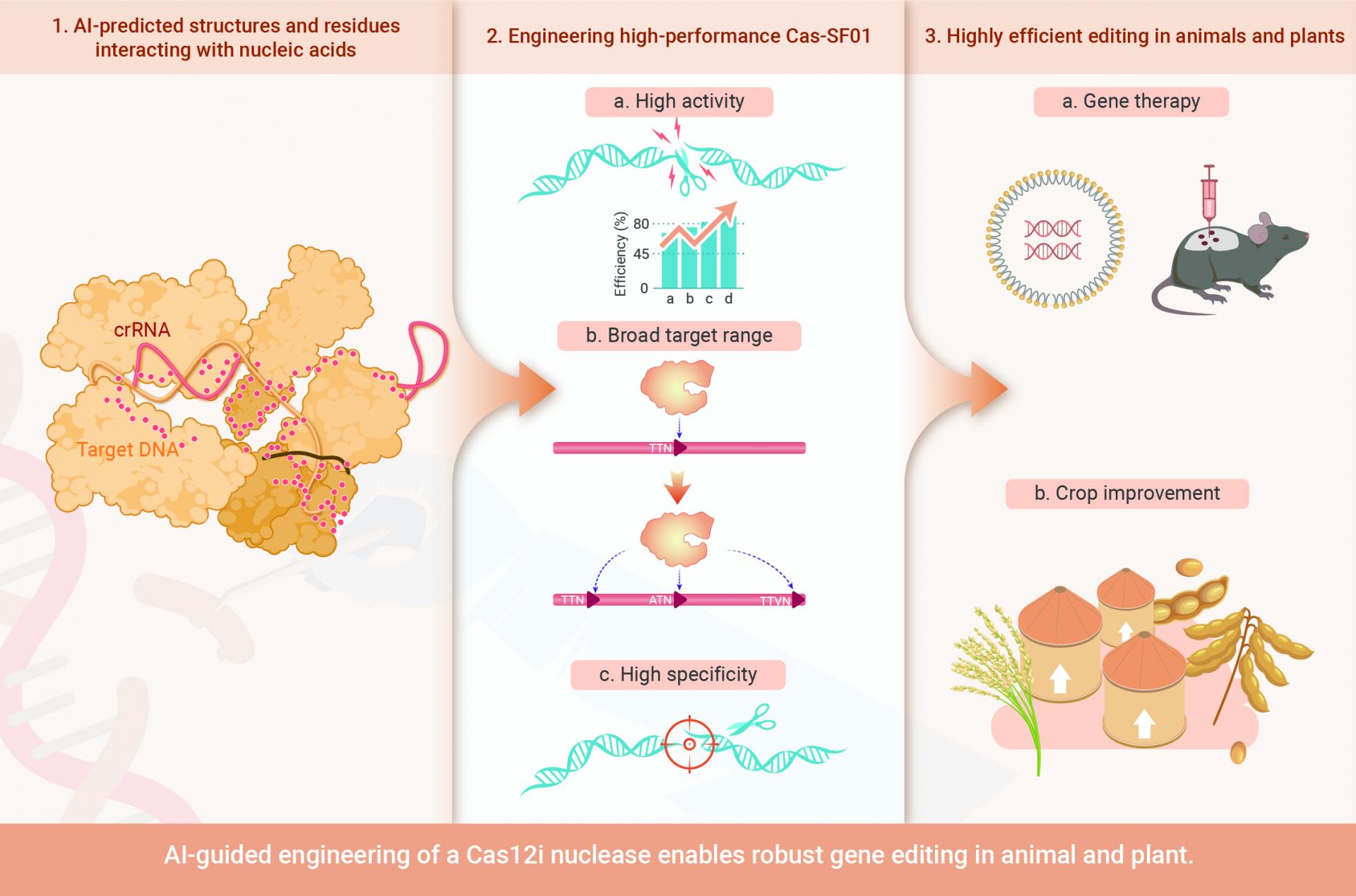
图1 Cas12i 活性改造策略
在本研究中,研究人员首先利用AlphaFold2预测了Cas12i3的三维结构,并将该结构与同家族的Cas12i1和Cas12i2蛋白结构进行比对,预测Cas12i3蛋白与核酸互作的氨基酸位点。通过对预测位点的突变建立一个150个点突变文库,研究人员利用荧光报告系统筛选到26个活性提升到1.5倍以上的Cas12i3变体。进一步通过对Cas12i3的REC结构域和WED结构域的点突进行组合并进行编辑效率分析,研究人员获得了活性提升约4倍的Cas-SF01(S7R/D233R/D267R/N369R/S433R)。与普遍使用的SpCas9和Cas12a蛋白相比,Cas-SF01在多个内源靶点的编辑效率超过了普遍使用的SpCas9和Cas12a,而在PAM识别方面,Cas-SF01展现出对TTN以外ATN及TTVN 的识别。此外,通过引入RuvC-II结构域的D876R单点突变,研究人员进一步获得了特异性显著提高的高保真版本Cas-SF01HiFi(图2)。
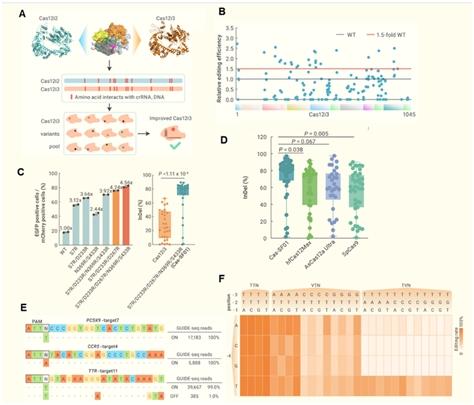
图2基于AI预测结构的高活性Cas-SF01设计、筛选组合、活性及PAM扩展测试
结合LNP脂质体递送系统,通过体内靶向小鼠的Ttr基因,Cas-SF01实现了与Cas9相当的编辑效率。在单子叶植物水稻多位点的稳定转化株系中,优化版本Cas-SF01显著提高编辑效率,达到75%左右。在双子叶植物辣椒和大豆的发根系统中,自然版本的Cas12i3未能检测到编辑事件,而Cas-SF01则表现出50%左右的编辑效率。以上数据表明Cas-SF01在动物及单双子叶植物中都能高效地编辑目的基因(图3)。综合而言,此项工作开发出了高效特异的基因编辑工具Cas-SF01,为将来的动植物基因编辑和基因治疗提供高效平台。
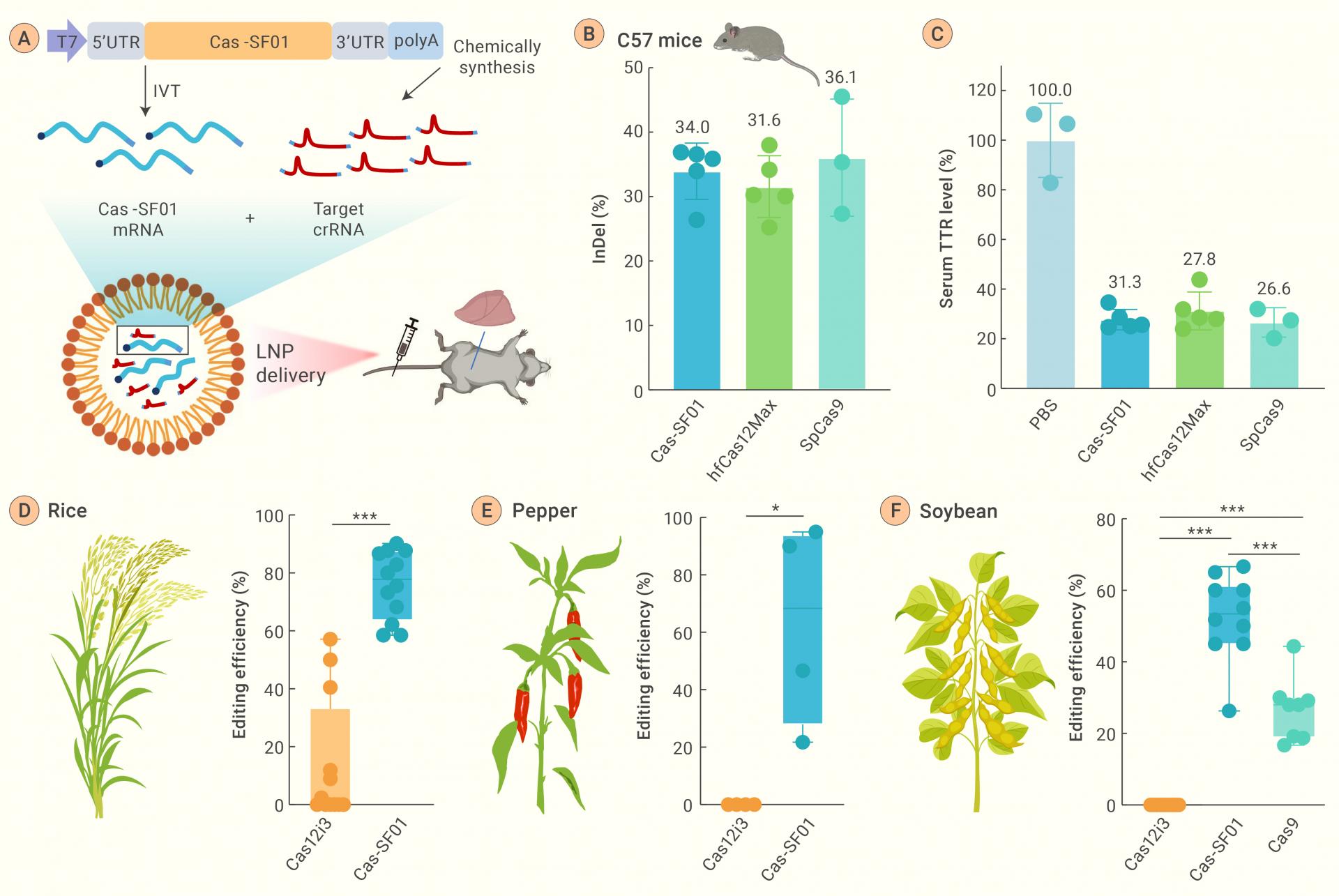
图3 Cas-SF01在动植物中都有高效的基因编辑效率
南方科技大学前沿生物技术研究院博士后段志强和访问学者梁亚峰为本论文的共同第一作者,南方科技大学前沿生物技术研究院博士后孙加雷、研究助理教授洪叶淳、医学院助理教授田瑞琳等参与本研究工作并提供重要的实验支持。南方科技大学前沿生物技术研究院院长朱健康院士为论文的通讯作者。南科大为论文第一单位。本项工作得到国家自然科学基金和山东舜丰生物科技有限公司等支持。
论文链接:https://www.cell.com/the-innovation/fulltext/S2666-6758(24)00002-X

